Bakterien sind wahre Überlebenskünstler, wenn es darum geht, sich an die unterschiedlichsten Lebensbedingungen anzupassen. Jeden noch so unwirklichen Lebensraum haben sie besiedelt. Sie können in heißen Quellen genauso gut überleben wie in den Eiswüsten der Polkappen. Ihr Überleben sichern sie wie alle Lebewesen, indem sie ständig Proteine herstellen, die sie für den Aufbau von Zellstrukturen benötigen oder die als Enzyme für die verschiedensten Stoffwechselreaktionen benötigt werden.
Worum gehts?
Besprochen werden die drei Phasen der bakterielle Transkription: Initiation – Elongation – Termination.
Für das Erkennen des Genortes ist das Sigma (σ) – Protein der RNA-Polymerase von Bedeutung. Was macht dieser Proteinanteil und wie funktioniert der Erkennungsmechanismus?
Woran erkennt die RNA-Polymerase, wann der Ablesevorgang zu beenden ist und wie verläuft dieser Mechanismus? Die
Polymerase – Sigma (σ) – Faktor
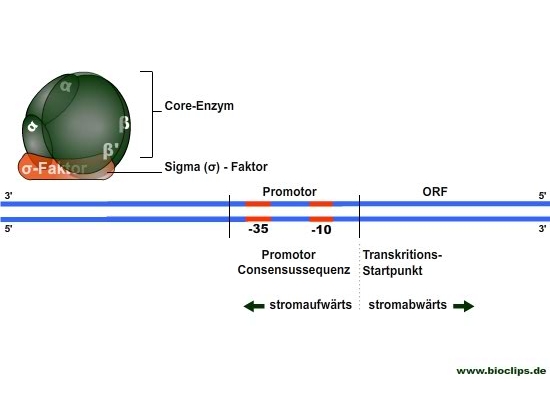
Die RNA-Polymerase besteht aus einer Core-Polymerase und einem Sigma (σ) -Faktor (-Protein). Core- und Sigma (σ) -Protein bilden ein Holoenzym.
Das Core-Protein, ist ein Proteinkomplex, der alles Notwendige für die Verlängerung der RNA-Kette enthält. Es besteht aus zwei α-Proteinen, einem β – und einem β‘-Protein.
Das Sigma (σ) -Protein wird ausschließlich für die Initiation – Start der Transkription – benötigt. Es kontrolliert die spezifische Bindung am Promotor und bewirkt die Spaltung der Doppelhelix.
Die Bedeutung des Sigma (σ) -Faktors
Bakterien enthalten verschiedene Sigma (σ) – Faktoren. Sie ermöglichen der Core-Polymerase zwischen unterschiedlichen Promotor-Sequenzen (Promotor Consensussequenz) zu unterscheiden. Man könnte auch sagen, der Sigma (σ) -Faktor ist der „Schlüssel“ für spezifische Promotor-Sequenzen.
Transkription
Initiation
- Das RNA-Polymerase-Holoenzym sucht die DNA nach Promotor-Sequenzen ab. Das Enzym bindet schwach an beliebigen Stellen auf der DNA und löst sich wieder, bis sie auf einen Promotor trifft.
- Die Bindung an die Promotorsequenz führt zur Ausbildung eines geschlossenen Komplexes. Das Holoenzym bildet hier mit der DNA einen schwach, aber dauerhaft gebundenen, geschlossenen Komplex, in dem die DNA noch doppelsträngig vorliegt.
- Für den Transkriptionsstart muss der geschlossene Komplex in einen offenen Komplex übergehen, indem die DNA entwunden wird, sodass die Basen ungepaart vorliegen. Durch Ausbildung des offenen Komplexes bindet die RNA-Polymerase fest an der DNA. Für die Entwindung unabdingbar ist auch der Sigma (σ) -Faktor.
- Die RNA-Polymerase entwindet nun weiter die DNA und beginnt somit unter Verwendung von Ribonucleosidtriphosphaten mit der RNA-Synthese.
- Der Sigma (σ) -Faktor verlässt den Komplex.
Elongation
- Bei der Elongation erfolgt die schrittweise Anlagerung von Ribonucleotiden unter Verwendung von Nucleosidtriosephosphaten.
- Nach der Verknüpfung von 9 Nucleotidbasen, verlässt der Sigma (σ) -Faktor den Transkriptionskomplex.
- Die Core-RNA-Polymerase liest den Matrizenstrang (Antisense-Strang) weiter schrittweise ab. Die Synthese der mRNA erfolgt mit einer Transkriptionsrate von etwa 45 Basen pro Sekunde. Durch die Entwindung entsteht eine Transkriptionsblase.
- Die Elongation erfolgt bis zum Terminationspunkt.
Termination
Am Ende des Gens bewirken spezifische Sequenzen die Freisetzung der RNA-Polymerase und der m-RNA. Die Transkription wird beendet. Diesen Vorgang nennt man Termination, wobei diese bei Bakterien nach zwei bekannten Mustern ablaufen kann.
Rho-abhängige Termination
Diese Termination ist von einem Protein mit dem Nahmen Rho abhängig. Das Rho-Protein erkennt eine spezifische Sequenz der mRNA, die G; C-reich ist und keine Sekundärstruktur ausbildet. Sie befindet sich hinter dem offenen Leseraster (ORF) und liegt damit hinter dem Stopp-Codon für die Translation (Translationsstopp). Diese sogenannte Transkriptionspause-Struktur verlangsamt oder stoppt den Ablesevorgang der RNA-Polymerase.
- Das Rho-Protein erkennt spezifische Sequenzen der mRNA (Transkriptionspausestruktur).
- Rho – Monomere verknüpfen sich in diesem Bereich mit der mRNA und bilden ein Hexamer, das Rho-Protein.
- Durch eine rotierende Bewegung (ATP-Verbrauch) zieht sich das Rho-Protein an die Polymerase heran, indem es die RNA aufwickelt.
- Kommt es zum Kontakt zwischen Rho und Polymerase wird der RNA-DNA-Hybrid durch die Helikaseaktivität des Rho-Proteins gelöst. Die Polymerase spaltet sich ab, die m-RNA wird freigesetzt.
Rho-unabhängige Termination
An der Rho-unabhängigen Termination ist weder der Rho-Faktor noch sind andere Cofaktoren beteiligt. Diese Termination erfordert eine G-C-reiche RNA-Sequenz, die komplementäre Basen enthält.
- Die G-C-reiche Sequenz bildet eine Schleife (komplementäre Basenpaarung) aus. Dabei handelt es sich um die Haarnadelstruktur.
- Die Haarnadel „erfasst“ die RNA-Polymerase und zwingt sie zum Anhalten.
- Der Kontakt zwischen Haarnadel und RNA-Polymerase führt zur Termination: Polymerase und Transkript werden freigesetzt.
 Punktlandung
Punktlandung
- Die RNA-Polymerase (Holoenzym) besteht aus einer Core-Polymerase und einem Sigma (σ) -Faktor. Die RNA-Polymerase bewirkt die Transkription am Matrizenstrang (Antisense-Strang).
- Bei der Initiation bindet die Polymerase mittels Sigma (σ) -Faktor an spezifischen Sequenzen des Promotors. Es entsteht ein geschlossener Komplex, der wiederum durch das Wirken des Sigma (σ) -Faktor in einen offenen Komplex (Transkriptions-Blase) übergeht.
- Nach Beendigung der Initiation verlässt der Sigma (σ) -Faktor den Initiationskomplex und die Core-Polymerase beginnt mit dem Ablesevorgang (Elongation).
- Bei Bakterien gibt zwei bekannte Terminationsmechanismen. Die Termination wird durch spezifische RNA-Sequenzen ausgelöst.
