Gene sind DNA-Abschnitte, die in RNA transkribiert werden: Genexpression durch Transkription. In den meisten Fällen enthalten RNA-Transkripte in ihren Nucleotidbasensequenzen die Information für die unterschiedlichsten Proteine. Wenn das so ist, handelt es sich bei den abgelesenen Genen um Strukturgene. Damit die Transkription und Translation fehlerfrei ablaufen, gibt es neben der Information für den Proteinaufbau auch DNA-Sequenzen, die signalgebend auf die Proteinsynthese einwirken.
Worum gehts?
Wie ist ein Bakteriengen aufgebaut?
Welche Funktion haben die Genabschnitte?
Was ist ein offenes Leseraster (ORF)?
Wie findet ein Ribosom das richtige offene Leseraster? Die
Genstruktur
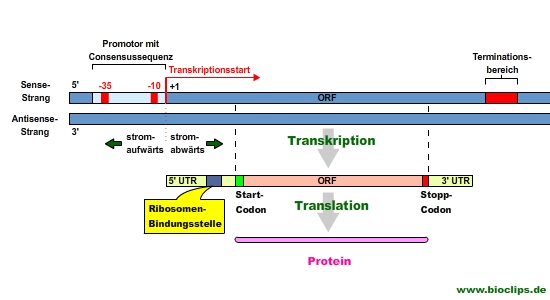
Das Bakterien-Gen kann man grundsätzlich in drei Abschnitte gliedern:
- Der Promotor ist die DNA-Region, die die Transkription kontrolliert.
- DNA-Abschnitt, der ab dem Transkriptionsstart in RNA übersetzt (transkribiert) wird.
- Terminationsbereich, ist der DNA-Abschnitt, welcher das Ende der Transkription bewirkt.
Der Promotor
- liegt stromaufwärts vom Transkriptionsstart vom codierenden Abschnitt
- besitzt zwei Haupterkennungsstellen, die -10 und -35 Region, die Angaben beziehen sich auf die Position stromaufwärts zum Transkriptionsstart (Nucleotide, die sich stromaufwärts vom Transkriptionsstart befinden, haben ein negatives Vorzeichen)
- die Haupterkennungsstellen werden von der RNA-Polymerase erkannt
- die Nucleotidbasensequenz der Haupterkennungsstellen können variieren
- Die Consensussequenz für die meisten Gene hat die Sequenzen TATAA (-10) sowie TTGACAT (-35). Die Consensussequenz erhält man durch den Vergleich vieler Promotoren, die in den entsprechenden Sequenzen weitestgehend übereinstimmen.
- eine hohe Übereinstimmung mit der Consensussequenz bewirkt ein permanentes Ablesen des Gens. Promotoren mit Abweichungen von der Consensussequenz benötigen Aktivatorproteine, um die Transkription in Gang zu setzen.
DNA-Abschnitt, der in RNA transkribiert wird
- Dieser DNA-Abschnitt beginnt mit dem Transkriptionsstart. Dabei handelt es sich um die erste Nucleotidbase (+1), die in RNA transkribiert wird. Ab diesem Punkt geht es „stromabwärts“, die durchnummerierten Nucleotidbasen erhalten ein positives Vorzeichen.
- In diesem Abschnitt befindet sich die Nucleotidbasensequenz, die für ein Protein codiert. Dieser Bereich ist das sogenannte offene Leseraster (engl. open reading frame, ORF).
- Das ORF wird durch Bereiche flankiert, die nicht translatiert werden. Vor dem ORF befindet sich die 5‘ untranslatierte Region (engl. untranslated region, 5´ UTR). Nach dem ORF lagert sich die 3‘ untranslatierte Region (3‘ UTR) an.
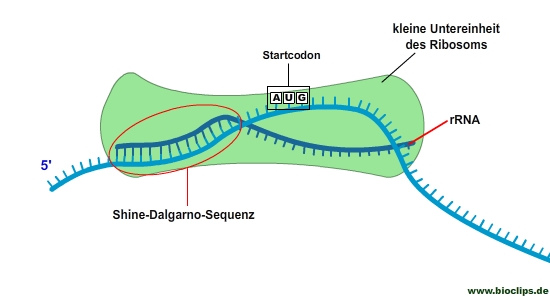
- Die 5‘ UTR enthält die Ribosomenbindungsstelle (Shine-Dalgarno-Sequenz), die für eine präzise Verbindung zwischen mRNA und Ribosom sorgt. Damit findet das Ribosom sehr genau das offene Leseraster, das Startcodon wird exakt in der P-Stelle ausgerichtet.
Terminationsbereich
- DNA-Sequenzen, die die Transkription anhalten.
Sense- und Antisense-Strang
Im DNA-Doppelstrang unterscheidet man noch in Sense-Strang und Anti-Sense-Strang.
Der Sense-Strang enthält die Sequenz für das Strukturgen und die entsprechenden Translationssignale, das heißt, die Nucleotidbasensequenz des Sense-Stranges entspricht der Nucleotidbasensequenz der mRNA bis auf den Unterschied, dass die RNA statt Thymin Uracil enthält.
Der Anti-Sense-Strang ist der Matrizenstrang, die „Druckvorlage“, die während der Transkription abgelesen wird. Das Ableseergebnis entspricht der Sense-Strang-Sequenz (Uracil ersetzt Thymin; „Spielerwechsel auf der gleichen Position“).
