Bakterien, die in heißen Quellen leben, können sich in weniger als 15 Minuten teilen. Damit erreichen sie in kürzester Zeit eine hohe Vermehrungsrate. Gleiches gilt u.a. für bakterielle Krankheitserreger wie Salmonellen, die sich unter den günstigen Bedingungen des menschlichen Verdauungssystems sehr schnell vermehren. Diese Massenvermehrung sichert das Überleben. Dazu benötigen Bakterien einen äußerst effizienten Replikationsmechanismus, der es ihnen ermöglicht, diese für das Überleben so wichtige Teilungsrate zu erreichen.
![]() Bei der bakteriellen Replikation sind ungefähr 20 verschiedene Komponenten beteiligt, die bei E. coli 4 639 221 Basenpaaren ablesen und verdoppeln. Die meisten sind in der Regel nicht Gegenstand des Biologieunterrichts in der Abiturstufe.
Bei der bakteriellen Replikation sind ungefähr 20 verschiedene Komponenten beteiligt, die bei E. coli 4 639 221 Basenpaaren ablesen und verdoppeln. Die meisten sind in der Regel nicht Gegenstand des Biologieunterrichts in der Abiturstufe.
 Worum gehts?
Worum gehts?
- In welche Phasen lässt sich der Replikationsvorgang gliedern und wie verläuft er bzw. was geschieht hier?
- Welche Zellbestandteile sind an der Replikation des Bakterienchromosoms beteiligt? Was ist ihre Funktion?
- Wie funktioniert der Replikationsstart (Initiation)?
- Wie verläuft die semikonservative Replikation des Bakterienchromosoms (Elongation)?
- Wie wird die Replikation abgeschlossen (Termination)?
![]() Kenntnisse über den Replikationsmechanismus bei Bakterien sind bedeutsam, um zu verstehen, wie bestimmte Antibiotika wirken oder wie die Polymerasekettenreaktion funktioniert. Einige beteiligte Moleküle haben Entsprechungen in der menschlichen Zelle. Gendefekte können hier die Ursache für Erbkrankheiten wie Xeroderma pigmentosum sein. Auch das Entstehen von Krebszellen lässt sich auf Fehler während der Replikation zurückführen.
Kenntnisse über den Replikationsmechanismus bei Bakterien sind bedeutsam, um zu verstehen, wie bestimmte Antibiotika wirken oder wie die Polymerasekettenreaktion funktioniert. Einige beteiligte Moleküle haben Entsprechungen in der menschlichen Zelle. Gendefekte können hier die Ursache für Erbkrankheiten wie Xeroderma pigmentosum sein. Auch das Entstehen von Krebszellen lässt sich auf Fehler während der Replikation zurückführen.
Der Replikationsmechanismus ist bidirektional

Der Reißverschluss meines Reisekoffers hat zwei Schieber. Zum Öffnen bewege ich beide Schieber in die entgegengesetzte Richtung. Eine „Replikationsblase“ entsteht. Geöffnet wurde bidirektional.
- Die Replikation der Bakterien-DNA beginnt an einem Startpunkt (Replikationsursprung oder einfach Ursprung).
- Ähnlich wie bei einem Reisekoffer öffnen Helikasen von diesem Ursprung ausgehend die Doppelhelix. Es entstehen zwei Replikationsgabeln.
- Wie die Reißverschlussschieber beim Koffer arbeiten die Helikasen in die entgegengesetzte Richtung (bidirektionale Replikation des Bakterienchromosoms), um dann an einem definierten Punkt aufeinander zu treffen (Terminationspunkt). Hier endet die Replikation.
- Die Einzelstränge dienen als Matrizen, die durch Anlagerung von Nucleotiden zu einer neuen Doppelhelix vervollständigt werden (semikonservative Replikation).
- Aufgrund der Wirkungsspezifität der DNA-Polymerasen kann die Synthese am Leitstrang fortlaufend (kontinuierlich) erfolgen. Am Folgestrang wird die DNA in kurzen Abschnitten (Okazaki-Fragmente) ergänzt (diskontinuierliche Replikation).
Die Phasen der Replikation
Initiation
- Initiationskomplex (DnaA-ATP-Protein lagert sich an).
- Doppelhelix wird entwunden.
- Anlagerung der DNA-Polymerasen.
Elongation
- Verlängerung der DNA-Einzelstränge durch Anlagerung von Nucleotiden
- Bereits während der Neusynthese wird Korrektur gelesen.
- Am Leitstrang erfolgt die Synthese kontinuierlich, am Folgestrang diskontinuierlich.
- Bei der diskontinuierlichen Replikation entstehen DNA-Fragment (Okazaki-Fragmente), die durch verschiedene Enzyme miteinander verbunden werden.
Termination
- Die Termination erfolgt am Terminationspunkt, der dem Ursprung gegenüber liegt.
- Die Enzyme werden entfernt. Es entstehen zwei Tochterchromosome, die als Ringe miteinander verkettet sind. Überschneidungspunkte sind Catenane.
- Die Verdopplung wird abgeschlossen.
Molekulare Strukturen
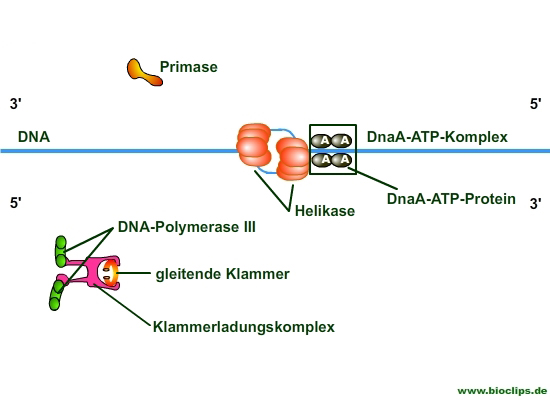
DNA-ATP-Komplex
- DnaA-Protein ist das Initiatorprotein, es erkennt spezifische Nukleotidsequenzen im Ursprung (oriC).
- DnaA bindet in Verbindung mit ATP.
- Die Bindung von DnaA-ATP unterstützt die Auftrennung der Doppelhelix (Replikationsblase).
Primase
- Durch die Primase erfolgt die Anlagerung von RNA-Primern, die mit ihrer freien OH-Gruppe die Starter-Sequenz für die DNA-Polymerase bilden.
Helikase
- Die Helikase bildet das Leitprotein der Replikationsmaschine.
- Es trennt unter ATP-Verbrauch die Doppelhelix in zwei Einzelstränge.
DNA-Polymerase III
- DNA-Polymerasen lagern neue Nukleotide komplementär an den Matrizenstrang an: Neusynthese des Einzelstrangs.
- Um sich anzulagern und die Neusynthese aufnehmen zu können, benötigt sie einen Primer mit einer freien 3′ OH-Gruppe.
- Gleichzeitig kontrolliert sie die synthetisierte DNA auf Fehler und korrigiert diese (Korrekturlesefunktion)
Gleitende Klammer
- Die Klammer fixiert die DNA-Polymerase an der DNA. Ohne sie würde sich die Polymerase von der DNA lösen.
Klammerladungskomplex
- Der Ladungskomplex besteht aus mehreren Proteinen und platziert die Klammer sowie die Polymerasen auf der DNA.
Initiation
- DnaA-ATP-Proteine binden innerhalb des Ursprungs.
- Die Bindung bewirkt die Strangtrennung.
- Die DNA-Helikasen lagern sich an den Ursprung an. Dazu benötigen sich jeweils einen Helikase-Ladungskomplex (Protein – im Film nicht dargestellt).
- Der Helikase-Ladungskomplex öffnet die Helikase und positioniert sie um den DNA-Einzelstrang.
- Die Helikasen binden Primasen, die Primer synthetisieren. Oben erfolgt vom Primer ausgehend die DNA-Synthese nach rechts unten nach links. In beiden Fällen handelt es sich um den Leitstrang (kontinuierliche Synthese).
- Der Klammerladungskomplex belädt die Primer der Leitstränge mit einer gleitenden Klammer.
- Die DNA-Polymerase bindet an der Klammer und synthetisiert am Leitstrang fortlaufend den neuen DNA-Einzelstrang.
- Die Helikasen arbeiten gegensätzlich gerichtet. Es lagern sich Primasen an, die Primer synthetisieren. Hier beginnt die diskontinuierliche Synthese am Folgestrang.
Elongation oder wie eine molekulare Maschine die DNA repliziert.
Nach der Initiation kann die DNA-Polymerase III am Leitstrang der Helikase kontinuierlich folgen. Die Neusynthese folgt der Laufrichtung der Helikase. Durch die Anlagerung neuer Nucleotide zur Synthese des neuen Einzelstranges steht der Polymerase immer eine frei 3′-OH Gruppe zur Verfügung (Die freie 3′-OH-Gruppe folgt der Helikase).
Durch die antiparallele Anordnung der DNA-Einzelstränge ergibt sich am unteren Einzelstrang bei der nach rechts laufenden Helikase das Problem, dass die Nucleotide immer mit ihrem 5′-Ende in Richtung Helikase zeigen. Es gibt aber keine Polymerasen, die am 5′-Ende ein neues Nucleotid anlagern können. Somit kann der neue Einzelstrang nur entgegen der Laufrichtung der Helikase wachsen.
Die Zelle löst dieses Problem auf sehr elegante Weise. Sie verbaut zwei DNA-III-Polymerasen, die jeweils für den oberen bzw. unteren Strang zuständig sind, zu einer „Replikationsmaschine“, das sogenannte Replisom. Zum Replisom gehören zudem die Helikase, die Primase sowie Einzelstrang-bindenden Proteine, die den Replikationsvorgang erleichtern (Auf sie wurde in der Animation verzichtet).
Das Replisom bildet einen stabilen Komplex, so dass beide Polymerasen der Helikase folgen. Durch die Fixierung des Einzelstranges über Polymerase und Gleitklammer steht der Polymerase am Folgestrang eine freie 3′-OH-Gruppe zur Anlagerung von Nucleotiden zur Verfügung. Da die Helikase sich weiterbewegt, bildet der freie Einzelstrang eine Schleife (loop). Durch diesen Trick kann die Einzelstrangsynthese entgegen der Laufrichtung der Helikase erfolgen.
Verlauf
- Die DNA-Polymerase III des oberen Stanges (Leitstrang) repliziert fortlaufend einen neuen DNA-Einzelstrang.
- Die DNA-Polymerase des Folgestranges synthetisiert am Folgestrang einen neuen Einzelstrang, der sich durch die Polymerase fädelt und zur Bildung der Schleife beiträgt.
- An der Helikase wird durch die Primase ein neuer Primer synthetisiert.
- Trifft die DNA-Polymerase auf einen zuvor synthetisierten Okazakifragment, wird sie freigesetzt und die Klammer entfernt.
- Eine neue Klammer wird auf einen freien Primer aufgesetzt.
- Die DNA-Polymerase bindet an diese Klammer und es beginnt die Synthese eines weiteren Okazaki-Fragments.
Das Problem der Okazaki-Fragmente
Die bei der diskontinuierlichen Replikation angelegten Okazaki-Fragmente müssen miteinander verbunden werden, um einen vollständigen Strang zu erzeugen. Wie das funktioniert, lässt sich hier nachlesen.
![]() Die Elongation kann als Modellvorstellung für die Replikation bei Eukaryontenzellen genutzt werden.
Die Elongation kann als Modellvorstellung für die Replikation bei Eukaryontenzellen genutzt werden.
Termination
Die Replikation wird am sogenannten Terminationspunkt beendet. Der Terminationspunkt liegt dem Ursprung gegenüber. Das Stoppsignal besteht aus 10 Terminationssequenzen. Treten die Polymerasen in diese Sequenzen ein, wirken bestimmte Proteine der Helikase entgegen. Anschließend werden die Polymerasen entfernt.
Nach der Replikation bilden die beiden Tochterstränge ein sogenanntes Catenan, das heißt, die beiden DNA-Ringe sind miteinander verkettet (Vergleiche Animation 1).
Die Catenan-Struktur wird beispielsweise durch verschiedene Enzyme aufgelöst, indem sie bestimmte Sequenzen erkennen und in diesen Abschnitten die Stränge spalten, um sie anschließend entsprechend zu verknüpfen.
Nach der Trennung wandern die Bakterienchromosomen zu den Zellpolen. Danach teilt sich die Zelle.
Bedeutung
Die bidirektionale Replikation ermögicht in kürzester Zeit die eine Verdopplung des Bakterienchromosoms und damit eine sehr kurze Generationsfolge. Das ist ein überlebenswichtiger Vorteil.
Dagegen müssen vielzellige Organismen ihre Replikation sehr genau kontrollieren, um unkontrolliertes Zellwachstum zu verhindern. Gerät die Zellteilung außer Kontrolle, entstehen Krebserkrankungen.
